摘要
背景
基于连锁不平衡(LD)的基因组 - 宽协会研究(GWAS)提供了有希望的用于检测和精细绘图的有希望的定量性状基因座(QTL)底层复杂的农艺性状。在这项研究中,我们探讨了在全球起源的224个弹簧大孔的各种集合中的特征标题,植物高度,千粒重,淀粉含量和粗蛋白质含量的变异基础。整个面板是基因分型,其使用Illumina的Goldengate技术含有1536个SNP的定制寡核苷酸池测定,导致957个成功的SNP覆盖所有染色体。形态特质“行类型”(两划线峰值与六划线钉)用于确认这种方法的高度选择性和灵敏度。本研究描述了GWA的上述农艺性状的QTL的检测。
结果
通过各种方法和六个亚组来调查小组中的人口结构,主要基于其尖峰形态和原产地。我们探讨了整个七种大麦染色体的整个面板中的联动不平衡(LD)的模式。观察到平均LD在临界水平以下衰减(R2-值0.2)在5-10厘米的地图距离内。面板内所有性状的表型变异都相当大。各性状的遗传力在0.90 ~ 0.95之间。对不同的统计模型进行了检验,以控制由种群结构引起的伪LD,并计算出其数量P -标记特征协会的价值。使用混合线性模型与血管关系进行控制,用于控制虚假的LD效果,我们发现总共有171个重要的标记特性关联,它描绘成107个QTL区域。这些特征遍布这些都可以分为57个新颖的QTL和50 QTL,这与以前映射的QTL位置一致。
结论
我们的结果表明,所描述的各种大麦面板可以有效地用于各种定量性状的GWA,条件是适当考虑群体结构。观察到的重要标记特质协会在大麦的重要农艺性状的遗传建筑方面提供了精致的洞察。然而,单个QTL仅用于一小部分的表型变异,这可能是由于标记覆盖率不足和/或在分析之前消除稀有等位基因。组合的SNP效应下降不阐述完整的表型方差可能支持定量性状的表达是由逃逸检测的大量非常小的影响引起的假设。尽管有这些限制,GWA的整合具有千分之连映射和越来越多的基因组序列信息,将促进农艺上重要基因的系统分离和随后分析其等位基因多样性。
背景
确定农艺性状的遗传基础一直是作物改良过程中面临的主要科学挑战之一。大多数重要的农艺性状都是数量性状,这使得识别潜在表型的遗传差异变得更加困难。连锁定位(analysis)是目前植物中检测复杂性状数量性状位点(QTL)最常用的方法。在连锁作图中,连锁不平衡是由两个亲本间的杂交建立群体而产生的。标记位点和表型性状的等位基因共分离使得连锁标记的鉴定成为可能。由于双亲本作图群体捕获的减数分裂事件数量有限,QTL作图的遗传分辨率往往局限在10-30 cM [1,2].此外,连锁分析只能在亲本起源的群体中取样一小部分可能的等位基因。
一种替代方法,关联映射(AM)称为LD映射依赖于现有的天然群体或设计的植物群体,以克服链接映射所固有的限制。LD映射利用人口中发生的祖先重组事件,并考虑了人口中存在的所有主要等位基因以鉴定显着的标记表型协会。LD映射首先在人类的遗传映射研究中引入[3.,4]最近被考虑用于植物研究。通过利用附近LOCI(LD)的等位基因的非随机关联,可以用一组映射标记舀出显着相关的基因组区域。映射的成功取决于表型数据的质量,人口规模和人口中存在的LD程度[5,6].一般来说,关联研究的力量取决于基因型标记和功能多态性之间的LD程度。不同物种的腐烂程度差异很大[7II)在一个物种和III内的不同群体中,也在给定的基因组中的不同基因座中[8,9].
LD定位基于两种策略:i)对选定的候选基因进行重新测序,ii)利用所有染色体的标记多态性进行全基因组关联[10].基因组协会研究(GWAs)在人类和动物遗传学的过去几年中越来越受欢迎和强大。出现更具成本效益的高通量基因分型平台的出现使得植物中的QTL映射的越来越有吸引力的方法[11].在过去的几年里,越来越多基于候选基因分析的关联研究已经发表(在[7])。这些包括:矮人[12[玉米中的植物合酶基因座[13[大麦的开花时间基因[14],这PsyI-人工智能小麦的基因座[15],这rhg-1大豆基因[16];和一系列候选基因拟南芥[17,18].
大麦(Hordeum Vulgare.)大约一万年前在新月沃土被驯化[19- - - - - -21].今天大麦是小麦,稻米和玉米后第四次最重要的谷物作物。除了其农业重要性外,大麦基因组还被认为是包括小麦和黑麦的Triticeae部落的其他作物种类的模型[22,23].在这方面,已开发出越来越多的大麦标记和序列资源,这些资源可以被有效利用[24- - - - - -26].在过去的几年中,候选基因基于大麦的研究[9,14,27].尽管这种方法的潜力已经在一些试点研究中得到了证明,但目前还没有对大麦进行常规标记覆盖的GWAS [28- - - - - -30.].
大麦等近交作物受非随机交配和后续选择的影响,具有较高的群体结构。这是典型的二棱和六棱大麦品种形成不同的亚群体,因为相应的育种程序依赖于不同的祖先。这同样适用于春大麦和冬大麦的亚群[31].由于面板中群体结构的混杂效应,AM中出现I型和II型错误的几率高于双亲本QTL分析[2,32- - - - - -34]提出了具体的统计方法,以考虑AM的人口结构[35].yu等人。[36]描述了比以前模型更好的混合线性模型(MLM)方法[37].但这些模型仍有各自的缺点,需要注意控制人口结构,平衡分析中的假阳性和假阴性率。
本研究的主要目的是利用GWAS分析春大麦抽穗期(HD)、株高(PHT)、千粒重(TGW)、淀粉含量(SC)和粗蛋白质含量(CPC)等复杂农艺性状的遗传多态性。我们研究了由Haseneyer等人先前描述的来自52个国家的224份春大麦收集。38].我们在人口结构和遗传多样性以及对GWA的影响方面提供全面的概述。为了研究七个大麦染色体的LD的动态,我们研究了LD衰变的模式。最后,我们识别并找到大量已知和新的QTL用于调查的特征。
方法
关联映射面板
关联映射面板由选自大麦核心集合(BCC)中选择的224个春季大麦广播组成[39以及在德国gatatersleben的IPK Genebank保存的大麦基因库。该小组由96个二排基因型和128个六排基因型组成,其中109份来自欧洲(EU), 45份来自西亚和北非(WANA), 40份来自东亚(EA)和30份来自美洲(AM)。大部分材料为改良品种(149份),部分材料为地方品种(57份)或育种系(18份)。有关种质资源的进一步信息可从欧洲大麦数据库(EBDB,http://barley.ipk-gatersleben.de/ebdb.php3).Haseneyer等人已经考虑了该面板和详细描述。[38].每一个入种都是单种子的后代,在温室条件下自交两代,随后在田间繁殖。
表型评价
在下列地点的2004年和2005年,斯图加特(德国西南部),Irlbach(Southteast Germany)和Wohde(德国北部),康复,饲养了25×15格设计。标题日期(HD)和工厂高度(PHT)在现场图中得分。千粒重(TGW)估计每绘制的采样颗粒。使用来自所有环境的地面种子样本的近红外反射光谱仪(NIR)估计淀粉含量(SC)和粗蛋白质含量(CPC)。为了将氮含量转化为粗蛋白值,我们认为是6.25的因子。我们遵循Naumann和Bassler中描述的方法[40]估计淀粉含量和氮含量。使用Genstat 9软件中实现的REML(剩余最大可能性)分析表型数据[41].通过将混合线性模型(MLM)拟合到多环境数据来计算方差分量。考虑到基因型方差百分比的所有特征估计了遗产,通过环境(E)方差和误差方差组分包括基因型(G)的总表型方差。表型平均蓝调(最佳线性无偏估计)估计考虑到GXE方差并用于结合研究。关于表型数据的更多信息可以从Hadeneyer等人获得。[38].
全基因组标记分析
针对SNP基因分型的DNA从八2周龄幼苗的膨胀叶样品中提取SNP基因分型。含有1536个等位基因特异性寡核苷酸的定制寡核苷酸池测定(IPK-OPA,UNPUMBL)用于通过Illumina的Goldengate Technology(Illumina,San Diego,CA)基因型基因型。IPK-opa主要建立在来自两种试验(Popa1,Popa2)的标记上的选择,这些标记是两种大麦品种“Barke”之间的多态,而且'morex'之间是多态性的。超过95%的IPK-OPA的IPK-OPA的SNP标记已包含在大麦共识图中[26].SNP基因分型是在加州大学(南加州基因分型联盟,UCLA)按照Fan等人的方案进行的。42,43].有关用于GWAS考虑的成功SNP标记的更多详细信息可作为补充信息(附加文件1:表S1)。
使用Illumina Beadstudio软件包对SNP数据进行评分(基因分型模块3.2.32;基因组浏览器3.2.9;Illumina,圣地亚哥,CA),可以处理原始杂交强度数据,从而聚类数据。在Beadstudio基因分型模块中实现的正常化过程包括异常值去除、背景校正和缩放。该算法使用贝叶斯模型将归一化强度值分配给三个可能的纯合子和杂合子基因型簇之一。严格的阈值评分(调用率> 0.9和绅士得分> 0.7)用于鉴别不明确的结果。未显示双组聚类的snp被严格排除在分析之外。在1536个SNP标记中,有985个标记产生了优质的基因型调用。在985个成功的SNP标记中,只有957个标记被遗传定位,我们使用这些标记进行分析(附加文件)1:表S1)。在224个基因型样本中,有12个基因型在检测中表现不佳(附加文件)2:表S2)。对于这12个基因型,超过90%的SNP标记数据缺失,因此被排除在后续分析之外。
基因型数据分析和群体结构
使用Powermarker 3.25计算每个SNP的多态信息含量(PIC)值。[44].主要等位基因频率(MAF)、次要等位基因频率(MAF)、基因多样性与Nei’s遗传距离(D.) [45计算和基于的NJ(邻居)树木(数据未显示)D.被计算了。从957 SNPS,使用大于0.05的MAF的918个SNP的最终组用于分析人口结构,LD和标记特征联想。
为了估计面板中的子组的数量,使用不同的方法和不同的软件包,并比较以确定收集中适当的人口结构。对于面板中的组数的定量评估,使用软件包结构V 2.2中实现的基于模型的方法进行贝叶斯聚类分析[46,47].此方法使用多基因座基因型数据将个体分配给集群或组(K.),而不事先知道他们的群体亲和力,并假定哈迪-温伯格平衡的位点。该程序使用918个SNP标记K.-Values 1到15(假设的子组数),100000伯爵迭代后跟50000 MCMC(马尔可夫链蒙特卡罗)迭代,用于准确参数估计。要验证我们对每个独立运行执行的结果的一致性K..使用具有相关等位基因频率的混合物模型。通过绘制从结构获得的估计的似然值[LNP(d)]来确定最可能的组数量K..LNP(D)是观察到的基因型分布的日志可能性K.簇,是由STRUCTURE模拟的输出。的K.值最能基于最大化数据的日志概率的标准或换句话说,LNP(d)达到高原的值[46].SNP标记数据集的结构结果与使用一组Diversity Array Technology (DArT)标记的结构运行结果相一致(Pasam et al. unpubl,附加文件)3.:图S1)。在第二种方法中,使用DARwin (Diversity analysis and Representation for windows)进行基于不同矩阵的主坐标分析(PCoA) [48].在第三种方法中,构建了基于Nei的遗传距离矩阵的NJDendγram。使用不同方法的集合中的子结构进行了比较,最终K.利用STRUCTURE确定值。对于这个K.值时,从STRUCTURE运行中提取q矩阵(人口隶属度估计)。这个矩阵提供了每个子组中每个加入的估计隶属系数。
连锁不平衡分析
LD的大小影响GWAS所需标记的数量和定位的分辨率。在许多情况下,遗传多样性受遗传材料的人口统计和育种历史所导致的种群结构的影响。通过使用HAPLOVIEW对SNP标记进行配对比较,对小组和亚组进行全基因组LD分析[49].通过使用平方等位基因频率相关性估计LD(R.2)在基因座对之间[50].当何时被认为是重要的LDP<0.001,其余的R.2价值观不被视为信息性。通过HaploView对每种染色体产生的LD图来观察染色体染色体LD的图案和分布。研究面板中全基因组中的平均LD衰减,显着的染色体内R.2绘制标记物之间的遗传距离(cm)的值。使用GENSTAT擦拭窒息的二级黄土曲线[41].的临界值R.2被取消链接的平方根转换估计R.2值得到一个正态分布的随机变量,并将该分布的参数第95个百分位作为临界值R.2价值[32].链接R.2指的是R.2在标记基因座之间具有大于50cm或独立连接基团的遗传距离。
关联分析
采用不同的统计模型进行计算P -将每个标记与感兴趣的特征相关联的值以及占人口结构,以避免Tassel v.2.1的杂散关联(http://www.maizegenetics.net.).我们遵循了公式y =xβ+ m + zu + e,在哪里y是表现型值的响应载体,β是有关人口结构的固定效果的矢量,α是标记效应的固定效果的载体,你是共同祖先和血液的随机效应的矢量E.是残留的矢量。X可以是来自主成分分析(PCA)的Q矩阵或PC,米表示标记和处的基因型Z是一个身份矩阵。选择包括一般线性模型(GLM)和混合线性模型(MLM)的六种模型以测试标记物性 - 关联(MTA)。比较结果以确定我们分析的最佳模型。PCA用流苏进行。前十个重要的PC解释了所有标记物的累积方差的43%。使用Tassel的918 SNP标记计算关联模型中进一步用于群体校正的一对族关系矩阵,其进一步用于群体校正的一对关系矩阵。51].对以下模型进行了检验:i)朴素模型:无种群结构修正的GLM模型;ii) q -模型:采用q -矩阵作为种群结构校正的GLM;iii) p模型:GLM,用pc对人口结构进行校正;iv) qk模型:采用q -矩阵和k -矩阵对人口结构进行校正的传销;v) pk模型:采用pc和k矩阵对人口结构进行校正的传销;vi) k模型:采用k矩阵对人口结构进行校正的传销[35,36,52,53].通过关联映射重新映射所有SNP标记,以确定如此建议的面板的映射解析24].关键P -用于评估MTA的重要性的值是基于每个特征分别的假发现率(FDR)计算的[54],被发现是非常严格的。考虑到用于占人口结构的模型的严格性,大多数误报都是固有的控制。因此,我们认为Chan等人提出的一种更广泛的方法。[55]用于确定重要MTA的阈值水平。建议底部0.1个百分位数的分布P -值可以被认为是显着的,在我们的分析中,各个特征导致阈值水平为0.05至0.09。或者,作为两者之间的折衷方法接近任意阈值P -值0.03用于所有特征和所有型号。通过安排log10获得这种相当粗略的估计P -值按降序排列,曲线开始变平时的值作为阈值。使用GENSTAT重新分析所有性状的关联模型[41]检查任何差异。
结果
表型数据
对所有特征观察到大量的表型变异。根据从所有环境的数据派生的残差来识别数据中的异常值,并从进一步分析中删除。对于特质期望日期,由于在各个地区之间评分此特征的差异,从2004年开始的数据被排除在分析之外。REML计算方差分量。结果证实,基因型方差对于所有特征都很重要(P< 0.001)。GxE的交互作用也很显著(P< 0.001),但只占总方差的一小部分。遗传力在0.90 ~ 0.95之间,表明数据的稳健性和低误差率。表中列出了HD、PHT、TGW、SC和CPC性状在所有环境下的年平均值、范围和遗传力1以及它们的频率分布在附加文件中说明4:图S2。彼此之间的农艺性状表现出的相关性在表中概述2.SC和CPC呈高度相关(-0.7),其他性状呈中-弱相关。PHT与HD、SC、CPC相关性较弱。TGW与SC呈正相关,与CPC呈负相关。据报道,二排和六排基因型之间存在显著的表型差异。所有性状的平均值在两组之间有显著差异(附加文件5:表S3)。六棱大麦各性状的变异均大于二棱大麦。穗形态(双排和六排)对TGW表型变异的影响最大,而PHT的影响最大(附加文件)6:表S4)。
基因型手段的最佳线性无偏估计(蓝调)由固定的基因型效应计算,以利用无偏均衡的平均估计。使用最佳线性无偏见的预测器(Blups)不太适合,因为它会导致双缩减[56].因此,我们在进一步的分析中使用了蓝调。然而,两个估计之间的估算之间的比较揭示了很高的一致性,这是高秘度的直接后果(附加文件7:图S3)。
人口结构和遗传多样性
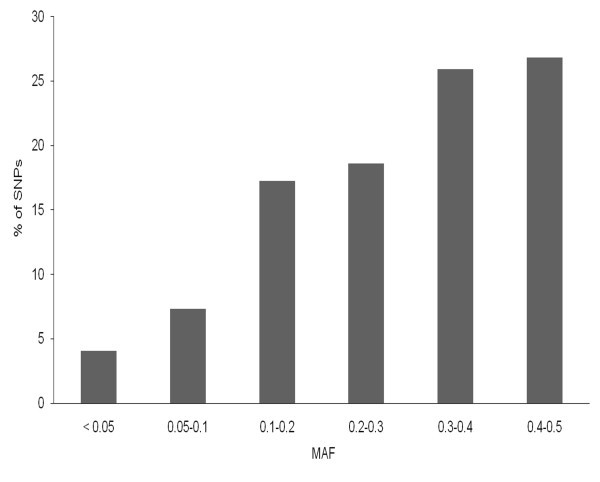
从高质量的985个SNP,957个标记已被遗传映射,因此考虑了这项研究。其中,由于MAF低于0.05,排除了39个SNP(4%)。剩下的SNP,大多数人揭示了MAF 0.1至0.5(图1).这些SNP标记分布在所有七条染色体上,平均间距为1.18厘米。SNP标记的分布并不完全均匀,染色体含有至少105个标记物在染色体4小时内和5小时最多164个标记物(表3.).为每个SNP计算的分集统计数据总结在附加文件中8:表S5。SNP的PIC值范围为0.09至0.5,平均为0.30。大多数标记(726)显示超过0.25的PIC值,展示了我们小组中这些标志的信息。每条染色体上标记的平均照片值范围为0.29(5h)至0.33(6h)。整个面板的平均基因多样性值为0.39,在0.09至0.5的范围内涂布。据报道,在几项研究中,大麦品种的分层与穗形态相辅调,主要是由于育种历史的结果[57,58].因此,我们分别为小组内的二棱和六棱大麦组以及六个亚组生成了类似的分子多样性统计数据。观察到的六棱大麦的平均PIC值(0.31)高于二棱大麦(0.27)。同样,六排遗传材料的平均基因多样性(0.38)高于两排遗传材料(0.33)。
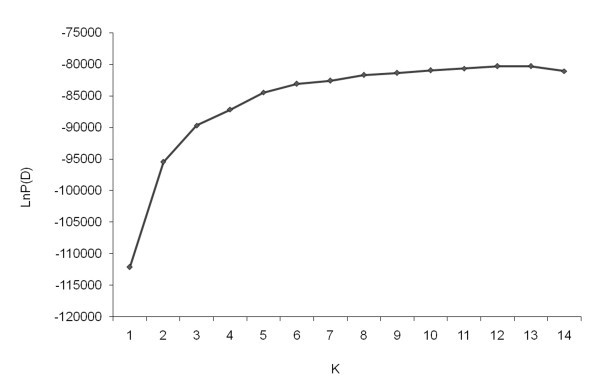
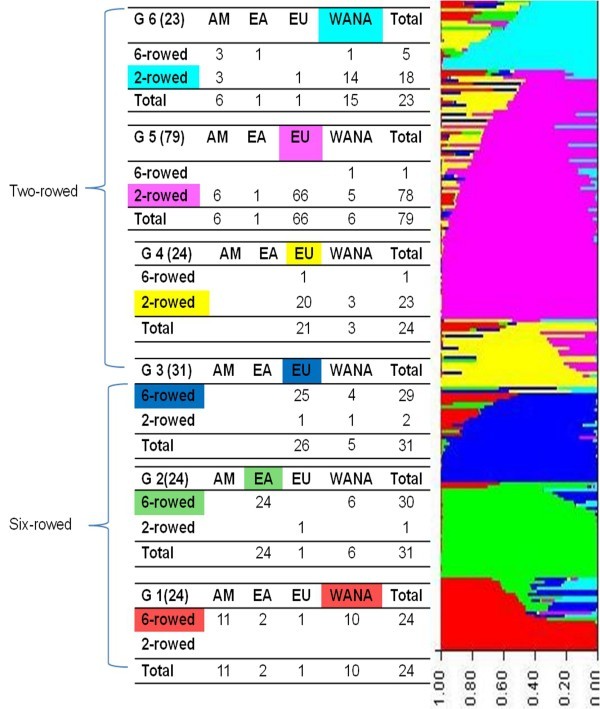
利用918个SNP标记和structure中基于模型的方法对212份大麦种质进行群体结构分析。LnP(D)似乎是一个递增函数K.对于所观察到的所有值。但是观察到LNP(d)的最显着增加的时候K.从1到2增加(图2).当K.==2小组明确分为两排和六划成的大型布勒,少数例外。由于LNP(d)值几乎达到高原,因此两种主要群体进一步分开了六个亚组。K.= 6。因此,我们选择的值为K.= 6对于我们的分析作为面板中存在的最小群体数量。不同的值K.仍有可能,但不会从质量上影响结果。如果估计至少50%的基因组信息属于一个组,则将一个加入组归为一个亚组。正如Haseneyer等人已经证明的那样,这些资源主要根据其穗状形态和地理来源聚集成群[38].六组定义为:第1组(G1):24个六划议的大师,主要来自AM和WANA;G2:31加入大多数六划出的大麦ea;G3:31加入来自欧盟的六划议六划痕;G4:24加入大多来自欧盟的两排;G5:79加入大多来自欧盟的两划大的仓库;G6:23加入从WANA和AM大多连续两排(图3.).PCOA证实了根据尖峰形态的群体的显性分层(附加档案9:图S4)和NJ DendOrgram(未示出)。在PCOA中,很明显,主要轴基于行类型分离加入,进一步分组与原点区域有关。总体而言,加入的聚类在各种方法中是一致的,我们进一步探讨了这些群体内的遗传多样性。每个组的摘要统计数据在表中报告了918个SNP标记4.观察到的基因多样性值范围为0.27,G1为0.27〜0.35;PI中的PIC值范围为0.22英寸G1的G5至0.29。成对遗传距离范围为0.006至0.628,总体平均值为0.39。已经计算了基团之间的平均总遗传距离,并且在G2(六圈,EA)和G5(两排欧盟)之间观察到0.36的最大遗传距离。同样地,发现G4(六划合,欧盟)和G5(六划合,欧盟)是密切相关的群体,平均遗传距离为0.17(表5).
连锁不平衡
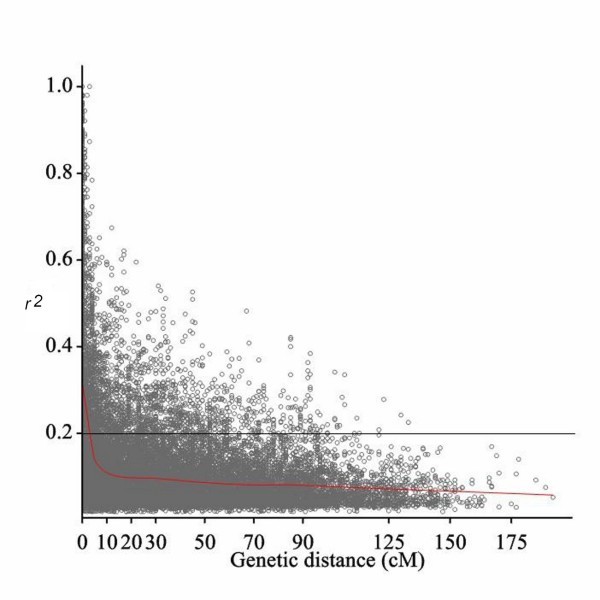
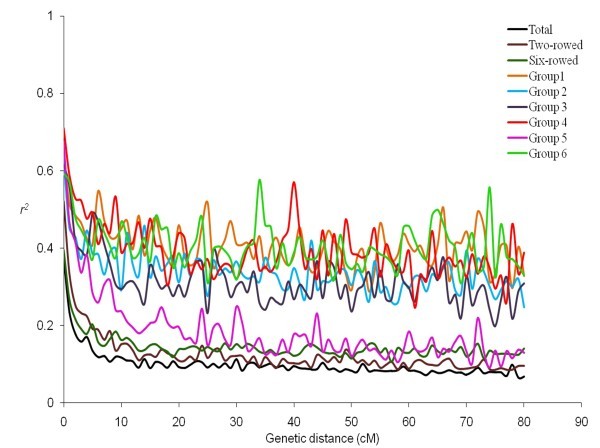
使用918个snp进行LD分析:i)全组,ii)二棱和六棱大麦,iii) 6个亚组。使用平方等位基因频率相关性(r2)并且被发现与遗传距离快速衰减。我们在小组中研究了LD的不同方面,观察到LD沿着染色体不同,其中高LD区域与低LD区域相互作用(附加文件10:图S5)。的临界值R.2,或基础LD,根据染色体间LD分析计算,估计为0.2,超过0.2则认为LD是由遗传连锁引起的。黄土曲线与临界值相交的点R.2被确定为人口的平均LD衰减。基于这些标准,染色体染色体的染色体LD衰减为5-10cm的单个染色体和整个基因组的平均LD衰减,为7cm(图4).大小的广泛变异R.2在给定的遗传距离处,检测到在染色体上的LD的范围内反映宽局部变化。之间的相关性R.2并发现标记距离是显着负面的(R.= -0.40)对于低于10厘米的标记,而具有较大距离的标记对显示出没有显着的相关性与R.2.
显着的染色体内R.2值(P< 0.001)的范围从0.02到1,整个面板的平均值为0.12。LD中所有显著位点中,13.7%的位点在临界位点以上R.2整个面板的值为0.2。根据标记间的遗传距离,对位点可分为4组:0 ~ 10 cM(紧密连锁标记)、11 ~ 20 cM(中度连锁标记)、21 ~ 50 cM(松散连锁标记)和> 50(独立标记)[59.].显著位点对的百分比和平均值R.2表中列出了整个小组和不同子小组中所有类别标记的值6.在所有基因座对中,只有39.4%的基因座在全组中出现显著LD。显著位点对的比例随位点间距离的增加而降低;62.2%的紧密连接标记显示显著性R.2.中度连锁标记中38.3%为松散连锁标记,独立标记中28.5%为显著LDR.2超过基础LD水平为0.2的值从一组紧密连接的标记物中的33.7%降低至10%,对于中等链接标记物,对于独立标记的4%小于4%。意思R.2对于未链接的标记对,值从0.2减少到0.08至0.08,以供不应的标记对。完整LD中的所有LOCI对都在遗传距离<5cm时间隔开。
亚组内联动不平衡模式
在染色体内水平,平均值R.2二棱和六棱大麦组的值在0.18 ~ 0.17之间,略高于平均值R.2整体面板。重要的百分比R.2除独立标记外,所有分类标记对的值在二排亚组均高于六排亚组。这一模式也类似于在基础水平0.2以上的LD值,与六棱类型组和整个面板相比,二棱大麦的LD衰减略慢。同样,的意思是R.2值估计的个体亚组,他们的范围从0.3 (G5)到0.49 (G4)。
子组中的LD衰减比整个面板慢得多。在图中5,垃圾R.2值是根据整个基因组的重组距离(cM)绘制的。在5 cM范围内,全组LD平均衰减在基值(0.2)以下,而二棱组和六棱组的基值在10-15 cM之间,且六棱组LD衰减速度快于二棱组。在G5内,LD在20 ~ 25 cM内衰减到基底层,其余亚群(G1、2、3、4、6)未达到基底层。各组平均LD衰减图呈现不同的模式。具体来说,在G4和G5亚组中,在距离45和74 cM处,我们观察到更大的LD峰。仔细研究这些峰值表明,这些区域的高LD是由低等位基因频率的标记引起的。个体亚群群体规模减少的结果是,单个等位基因的存在可能已经显示出MAF高于临界阈值。不同亚种群的LD衰变模式不同,可能反映了它们的繁殖历史[1]并且可能会影响面板的QTL映射分辨率。
协会委员会的评估
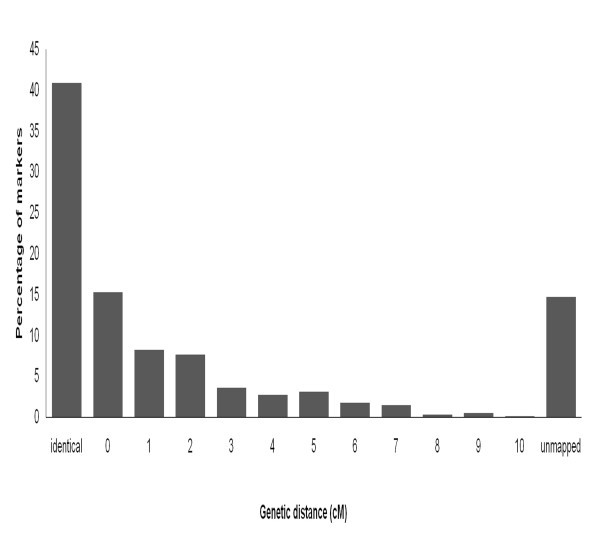
使用LD方法重新映射所有918个SNP。用于群体结构的亲属性核算模型用于验证标记的遗传地图位置。我们将每个标记信息用作单独的特征,并使用剩余的SNP进行分析,以找到最显着的关联标记。所讨论的目标标记与最高相关标记之间的地图距离用于评估面板的分辨率。超过85%的SNP标记的遗传地图位置在其原始地图位置的0-10厘米的距离内,其中大部分重新映射在同一位置(图6).标记的这种重新映射表明,在我们的面板中捕获的QTL的分辨率将在5-10厘米的范围内。
关联分析
模型的比较
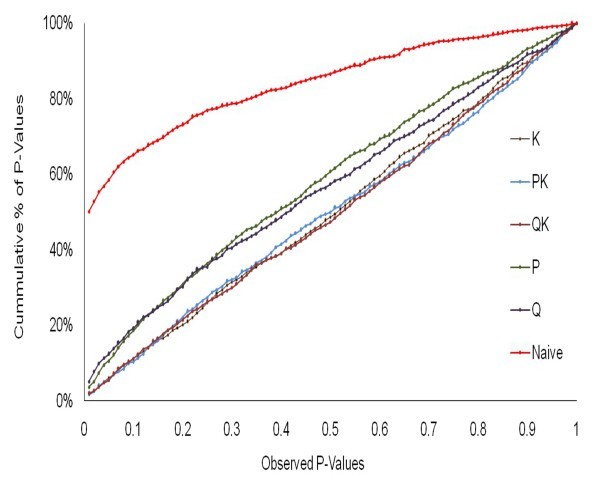
我们测试了几个模型来检测SNP标记和农艺性状之间的关联。由于我们小组中存在的复杂性和相当数量的人口结构,我们在使用单纯(简单)模型时观察到许多虚假的关联。因此,我们通过比较各种线性模型减少假阳性关联膨胀的能力来评估各种线性模型用于解释人口结构的有效性。为此排名P -通过使用尖峰形态作为表型特征,以累积方式绘制来自GWA的值(图7).正如康等人所证明的那样。[53]分布P -理想情况下,值应遵循均匀分布,与预期的偏差较小P -值。模型QK, PK, K显示了很好的适合P -值,而另一个模型的特点是超过小的P -价值观与丰富的虚假关联的丰富。在“天真”的模型的情况下,这尤其明显,其中近一半P-Values小于0.01。另一方面,K模型类似于PK和QK模型,在显示高度均匀的分布时P -值,同时需要更少的计算时间。不管模型如何,主要的标记性状关联不断被检测到。然而,模型越严格,检测到的虚假背景关联就越少。所有考虑GWAS的模型都是针对特征尖峰形态提出的(附加文件11:图S6)。对于所有其他性状,本文只介绍和讨论k模型的结果。
大麦尖峰形态(行类型)
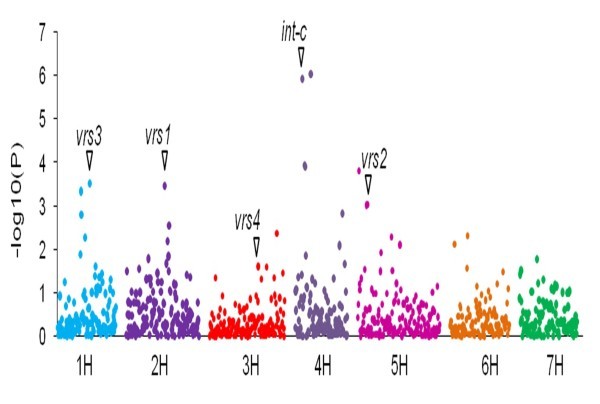
除了比较不同的AM模型外,我们的目的是检验刺突形态特征,作为GWAS概念的证明,并评估关联小组的分辨率。根据大麦穗形态,大麦可分为二棱型和六棱型,有关这一性状的基因已被很好地记录下来,其中一些已经克隆[29,30.,60.].我们在面板中打入了行类型字符,并考虑了所有型号的918个标记。当标志物的主要效应显着均为0.03 [-log时,考虑了标记性状关联。10(0.03) = 1.5)。通过k -模型,共得到34个与性状行类型显著相关的标记。(附加文件11:图S6)。结果与之前的行类型研究一致(见图8).
标题日期
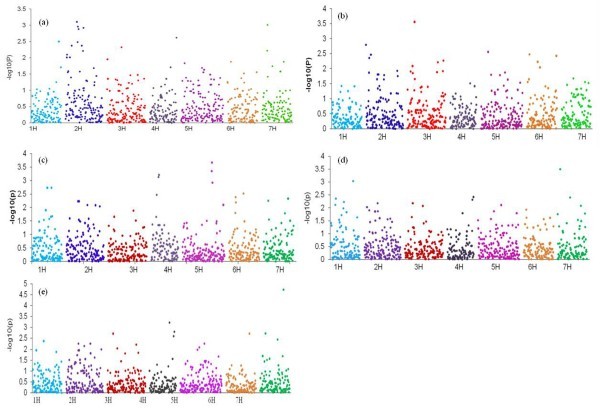
发现三十四个标记与标题日期(HD)显着相关。将这些分为19个QTL,位于所有染色体上。遗传距离在5-10cm的遗传距离内的显着标记性状关联被描绘成单个QTL。染色体2h竖琴与特征相关的最大标记数(图9).这些SNP标记的关联结果与之前定位的花期QTL的基因组区域有效对应。这些包括各种突出的开花途径基因的基因组区域Ppd-H1、HvFT1 HvCO1和HVCO3.(见表7).
植物高度
32个标记与株高显著相关。这些标记共检测到19个QTL(见表)8).除1H染色体外,所有染色体上均发现显著相关标记,多数位于2H和3H(图)9 b).
千粒重
36个标记产生21个与千粒重显著相关的QTL (TGW,图9 c).与该性状显著相关的标记均存在于所有染色体上。如预期的那样,一些与TGW相关的QTL与穗形态的QTL重叠。标记SNP56、SNP215、SNP385和SNP458共同定位于同一区域VRS3,VRS1,VRS4和Int-c基因组区域(表9).
淀粉含量
有35个标记与淀粉含量(SC)显著相关。这些标记共形成25个QTL(图)9 d).所有染色体上均存在淀粉含量显著相关的标记。类似于TGW标记对应的VRS3.区域(SNP56和SNP66)与淀粉含量显著相关。几个重要的标记,与之前定位的基因和SC的QTL共同定位(表10).
蛋白质含量
研究发现34个标记与粗蛋白质含量(CPC)显著相关。这些标记共检测到23个QTL(图)9 e),分布在所有染色体上。一些蛋白质含量的QTL与CPC的QTL区域重叠(见表)11).
讨论
在本研究中,我们描述了全基因组关联映射在各种春季大麦基因型面板中的应用,用于农艺性状。对于我们确定的每个分析的特征,我们识别了19到25 QTL。派生QTL位置的大部分是在各种千分之一映射群体中识别的QTL的一致性(表7,8,9,10,11).GWA受到表型数据的质量的强烈影响[79.].在目前的研究中,所有性状的遗传力超过0.9,表型手段反映了面板中广泛的变异。由于育种历史的不同和穗形态的多效性影响,二棱和六棱组观察到的差异是预料中的(附加文件5:表S3)。六棱组所有性状的表型变异均高于二棱组,这与该亚群较高的遗传多样性是一致的(表)4).通过对群体结构的进一步分析,发现了6个亚群,这些亚群主要由穗型形态和地理来源定义,这两个亚群对农艺性状的表达都有影响。
遗传多样性和人口结构
有争议的是,一个关联图谱应该足够表型和分子多样性的结果可靠的关联结果。由于有大量的可定位SNP标记,可以以多并行方式询问[26],我们能够达到高标记覆盖率,即每1.18 cM有1个标记。在这组材料中观察到的平均PIC(0.30)和基因多样性(0.33)值与之前使用双等位基因标记的研究结果具有可比性。不同的染色体和不同的种质亚群间的PIC值不同(见表)3.& 4).在所有染色体中,6h染色体的PIC值最高(0.33),这与Rostoks等人的观察结果一致[24在一组欧洲大麦栽培品种中。我们通过采用不同的方法(structure, PCoA和nj - dendrgram)确定了小组中的种群结构,并发现了相似的结果。之前的一些研究,如Maliysheva-Otto等人[57,罗斯托克斯等人[24],张等人。[58和Hamblin等人[80已经表明,生长习惯,穗形态和地理起源是镜像大麦人口结构的主要因素。由于目前的研究仅限于春麦,因此穗形态和地理起源是人口子结构的基本决定因素(G1至G6)(图3.).本面板中包含的55项Landrace载体分布在所有群体中。子组G1,G2和G3主要是六划线的布雷和亚组G4,G5和G6主要包括两划两划线的布雷。面板中的两排大师彼此密切相关,而不是六划议的六划线的六角形,这与张等人的研究结果相反。[58为加拿大种质资源。虽然在我们的讨论小组中,双排大麦的数量甚至超过了六排大麦,但它们多样性有限的原因可能是大多数源自欧洲。遗传资源的地理分布对在种群中抽样的等位基因的多样性有重大影响[57].在欧洲,两棱大麦主要是作为麦芽生产的原料种植的。麦芽品质是一个数量性状。在相应的育种计划中使用有限数量的主要祖先导致了遗传多样性的减少,并伴随形成了一个独特的亚群体,正如我们现在的小组所看到的[81.].
LD配置及结果
LD作图的分辨率取决于LD在整个基因组中的程度以及LD随遗传距离衰减的速率[82.,83.].在使用不同分子标记的各种群体中,诸如AFLP,SSR和DART(SSR和DART)的各种群体以前已经报道了基因组LD研究[57,58,84.,然而,很少有研究依赖于1000多个标记。在我们对世界范围春大麦种质资源的研究中,染色体内全基因组LD低于临界值R.2-值(0.2),遗传距离为5 cM。需要记住的是,这是一个平均值,总结了大量的染色体内LD变异。在我们的小组中,不同染色体的染色体内LD范围从5-10厘米不等,每条染色体的模式不同(附加文件)10:图S5)。以往的研究发现,不同大麦种群的枯叶腐烂程度不同[9,29,83.以及不同染色体之间的差异[24].在Comadran等人的研究中,LD的衰变更为迅速[85.可能是由于纳入集合中的体力。Caldwell等人。[9还表明,与精英大麦品种相比,LD在大麦地塑料中衰减更快。在我们的面板中发现了超过10厘米超过10厘米的广泛的LD,因为基础水平高于基础(33.7%)的大多数显着的LD值是由于紧密相关的标记。未解链标记的显着轨道间LD值(4%)可能是人口结构的结果(表6).我们发现了一些紧密联系的标记,这些标记处于完全联系平衡(LE),而一些远方的链接标记表现出高LD值。这反映了染色体沿着染色体的LD图案的动态变化,因为它在该面板中显示了几种转录因子的序列水平[27].至于个别的子组,其部分意义重大R.2- 在基础级(0.2)上方的值在六划度内比在这些组中指示高LD的两排组中更高。有趣的是,除了G5之外,所有子组都延伸超过30厘米,其中LD延伸到大约20-25厘米(图5).这很可能是因为G5比其他亚群的人口规模更大。在亚组中观察到的广泛LD可能是由于它们的种群规模减少和相关性增加。
全基因组关联映射
尽管GWA有优势来定位遗传多态性的基础农艺性状,但这种方法可能由于人口结构而遭受误报的膨胀[4,52,86.].在先前的研究中提出并测试了几种矫正人口结构效果的统计模型[37,52,87.].由于我们在本面板中发现了相当多的结构,我们使用线性模型来控制人口结构,并减少假阳性关联。类似于以前比较异种和自花授粉物种GWAS模型的研究[37,52],我们的结果表明k模型、QK模型和PK模型的表现优于其他模型(图7).此外,对于k模型,计算时间更快,不需要在面板中识别适当的人口结构(q矩阵)等额外步骤。由于在探索性分析中,三种方法的结果基本一致,所以采用k模型对所有性状进行完全分析,以避免数据冗余。然而,我们应该记住,修正人口结构不仅会减少假阳性的频率,而且在性格状态与人口结构密切相关的情况下,也可能导致假阴性[28].
为了确认使用可用标记范围的关联映射的面板的效率和分辨率,我们使用k模型重新映射所有918个SNP。从918 SNPS,783重新映射到其原始位置的10厘米范围内。只有14%的标记映射超过10厘米。在成功重新映射的标记中,超过95%标记的标记在距离原始地图位置距离5厘米的距离,指示我们面板的映射分辨率(见图6).罗斯托克斯等[24]已经使用同样的方法,用标记子集评估他们收集的大麦的GWAS,并成功地绘制了80%的标记。
为了展示面板和模型的GWAS的适用性,首先分析了尖峰形态(行类型)(图8).这个性状很容易被标记,并且从农学和驯化的角度来看是重要的。行型的遗传基础已经为人所知,已经定位了几个QTL,克隆了多个基因[29,60.,88.].我们发现这个性状有34个标记-性状关联(图)8).我们所确定的行类型的标记-性状关联与所有先前确定的主要位点并行vrs1[88.],vrs2、vrs3 vrs4和INT-C.[29,30.].为行类型检测到的其他,不太重要的关联可能与任何已知的主要基因座无关,并且需要进一步探索。行类型的结果是我们春季大麦面板中GWA的概念证明,反映了GWAS在近亲繁殖物种中的高分辨率QTL映射的效率。一些行类型QTL与其他特征的相关区域重叠,尤其是具有特征TGW,SC和CPC(附加文件12:图S7)。正如预期的那样,两排大麦的TGW比六划队类型更高,因为两排尖峰中的水槽器官(内核数量小于六划分的尖峰。虽然Spike Architects对TGW的影响显然是抗脂病,但它对SC和CPC的影响是育种历史和最终使用质量的结果。在麦芽大麦的情况下,品种通常为高淀粉和低蛋白质含量。在欧洲,大多数两划大麦是麦芽的优选,而六划的大麦主要用作饲料,其特征在于高蛋白质含量[22].因此,在我们的小组中,二排型的淀粉含量高于六排型,而蛋白质含量低于六排型5:表S3)。正如预期的那样,研究小组中包括的地方种族没有显示出这种分层,因为他们没有承受这种选择压力。
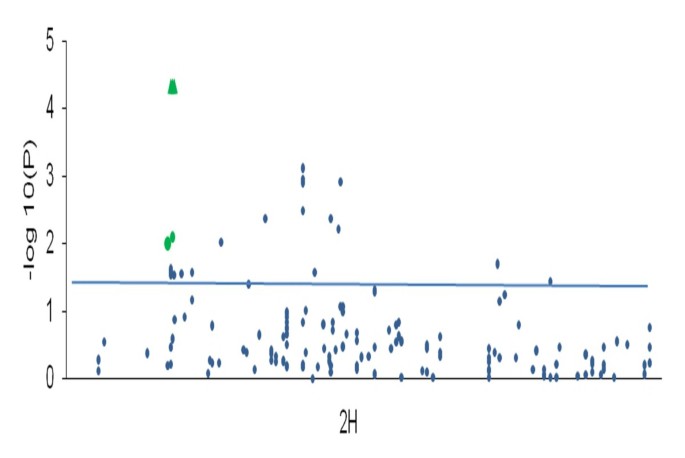
抽穗期(hed)反映了植物对环境的适应,是一个复杂的性状,受远系育种中众多QTL的影响[89.]和近亲繁殖的物种[61.].许多SNP标记被发现与HD性状相关(图9)我们报告共34个重要的SNP定义19 QTL。这些QTL中的一些据报道以港口主要基因的基因组区域为包括HvFT3, PpdH1, HvFT4, eps2, HvGI, HvCO3, HvFT1和HVCO1.(表7).在使用相同面板的先前研究中,从三个开花时间候选基因的片段重新测序,基因内的SNPPPDH1揭示了对HD的最大影响[14].在目前的Gwas,SNP位于附近(约2厘米)PPDH1与HD显示出重大关联(表7).通过进一步包括所有PPDH1来自Stracke等人的SNP。[14]进入我们的GWA,这些SNP揭示了所有使用的所有标记的最高关联(图10).这些发现者对假设提供了强度,即标记覆盖的进一步增加将导致检测额外的关联或改善现有QTL的意义。
2H染色体HD性状与IPK-OPA单核苷酸多态性的关联分析及重测序PPDH1片段[14].蓝色圆圈代表2H染色体上的IPK-OPA SNP,绿色圆圈代表与HD显着相关的IPK-OPA SNP,并附近PPDH1基因,绿色三角形代表重新测序的SNPPPDH1片段
在PHT性状上,我们发现了19个推测的QTL区域,分别位于2H、3H、4H、5H、6H和7H染色体上,包括32个标记性状关联。为了减少倒伏和提高收获指数,世界各地已经开发了半矮秆和矮秆品种。不同的基因/等位基因被部署在不同的地理区域:GA敏感sdw1侏儒基因已在美国和澳大利亚部署,而其等位基因形式被称为日本电装,经常在欧洲两排足的种质中看到。隐性uzu等位基因在日语,中韩品种中发现了[70,90.].对于PHT的许多QTL与先前映射的QTL和基因相一致(表8).2H染色体上的QTL4_PHT与的定位位置一致sdw3在赤霉素不敏感矮化大麦中起主要作用[68.].两种等位基因形式的矮化基因Denso / SDW1.映射到位于染色体长臂上的QTL8_PHT相同的基因组区域[70].QTL7_PHT距离大约10厘米距离uzu基于谷物基因数据库中呈现的共识图的基因座。
千粒重(TGW)是对最终产量直接影响的主要产量组分之一。对于这种特征来发现共同21个QTL,其中一些QTL在行类型基因附近。先前映射QTL在同一基因组区域中进一步确认了一些QTL(表9).观察到5HL上的QTL14_TGW以实现像PHT,SC和CPC等其他特征。
如上所述,淀粉和蛋白质的含量是最终使用质量的主要决定因素。检测到的25个淀粉含量QTL中,有几个与之前鉴定的QTL相一致(见表1)10).这些包括QTL与酸性洗涤剂纤维(ADF)含量,淀粉颗粒尺寸和颗粒形状等相关性状的QTL [76.].7H上的QTL21_SC位于蜡实已知编码轨迹颗粒结合淀粉合酶I(GBSS I.),催化淀粉糖的合成[91.,92.].籽粒粗蛋白质含量共鉴定到23个QTL,均位于7条染色体上。其中11个QTL与之前定位的QTL共定位,12个QTL是新定位的QTL11).有趣的是,SC和CPC的大部分QTL位于7H染色体上。部分SC的QTL与CPC的QTL一致,如染色体1H (55 cM)、2H (33.74 cM)、3H (55 cM)、5H (110 cM)和7H (12 cM和121 cM)10& 11)。由于这两个性状的负相关关系,QTL的一致性是可以预期的2).另一方面,我们也不能排除部分共享QTL是基础基因连锁的结果。
GWA仅揭示了小效果
即使在本研究中观察到的最佳协会也只显示了谦虚的r2对应的SNP的值(解释的遗传性状变化的百分比),暗示每个SNP预测的低方差。这是由QTL'QSCH7A'举例说明的,其在QTL映射研究中解释了SC中的47%的变化[76.].在本研究中,与Qsch7a位于同一基因组区域的'QTL23_SC'只能解释0.2%的变异。许多人类GWAS报告R较低2值和未解释的变化的其余变化被称为“无法解释的遗传性遗传性”[93.].Roy等人。[94.R2-值范围从0.2%到3.95%的GWAS植物,这与我们目前的结果很好。在一项关于“身高”特征的联合研究中,在严格的阈值下鉴定出了40个令人印象深刻的基因型变异。这些因素加在一起只能解释大约5%的人体身高变化[95.,96].对于这种“缺少遗传性”的可能解释包括I)标记覆盖不足,在因果多态性不具有基因分型SNP的完美LD的情况下减少了检测关联的力量和这种SNP标记解释的变化。这已经证明了本研究的影响PPDH1HD的基因;II)从分析中删除了主要效果的罕见等位基因(MAF <5%),并且在它们相关的情况下会出现未检测到的;III)性格或性状的表达取决于大量基因/ QTL,具有逃避统计检测的小个体效果;iv)可用于检测GWAS和V)偏见的统计方法的统计方法的不足2由于小组中的人口分层水平,为单独的SNP [93.,95.,97- - - - - -99].尽管上述原因主要在人类GWA的背景下讨论,但它们也涉及植物和其他生物的GWA。除了上述原因之外,用于分析的统计模型将影响SNP解释的变化。随着模型的严格性和阈值增加,将减少检测小效果SNP的功率。我们观察到,在使用针对GWA的严格模型的情况下,模型本身解释了特征变异的较大部分,并且留下了越少的变异来解释遗传效应。对于Trait HD The K模型,解释了特性的近70%的变化。减少模型的严格性将增加标记解释的变化,但同时会导致更苛刻的阳性。特别是在近亲繁殖的作物,如大麦,通过应用更简单的模型,难以完全妨碍基因型之间关系的影响。因此,Gwas在近亲繁殖作物的高度结构群体中,例如大麦的繁殖作物将取决于对灵敏度与选择性的模型的仔细优化。
结论
总体而言,我们的结果提供了关于GRWAS在近亲繁殖的作物等繁殖作物中的GWA的机会和陷阱的新细节。本研究的结果提供了对大麦(HD,PHT,TGW,SC和CPC)重要农艺性状的遗传建筑的洞察。总共确定了这些特征的107 QTL。一些基因组区域突出一个以上特征的QTL,并且根据地图比较,已发现50个QTL,以先前映射的QTL同意。对于所有特征在一起,已检测到57个新型QTL。为了减轻GWAS在近亲繁殖作物中,未来的协会研究可能会实施新的策略,如联合联系和LD映射,这些策略已经成功地应用于各种物种[89.,One hundred.- - - - - -102].此外,到精细的地图和“孟异化”所选的QTLS,对于大麦(品种,Landraces,Wild Barley)的不同Genepools,观察到的LD衰变的交错模式可以与初步映射和标记饱和策略相结合,利用了越来越多的基因组序列[30.,103].最近通过鉴定候选基因来证明这种方法的可行性花青素2使用关联映射的组合的基因座,然后在千分之一群中进行分离分析和BAC CONTIG分析[104].
参考资料
- 1.
Flint-Garcia SA,Thornsberry JM,Buckler ES:植物中连锁不平衡的结构。Annu Rev植物Biol。2003,54:357-374。
- 2.
朱C,Gore M,Buckler Es,Yu J:植物中关联映射的地位和展望。植物基因组杂志。2008,1(1):5-
- 3.
哈斯贝卡,德拉切pelle,凯蒂拉,西斯托宁,韦维安,等:芬兰孤立的奠基人群体的连锁不平衡图谱研究。自然遗传学。1992,2(3):204-211。
- 4.
着陆器ES,Schork NJ:复杂性状的遗传解剖。科学。1994,265(5181):2037-2048。
- 5.
flet - garcia SA, Thuillet AC, Yu J, Pressoir G, Romero SM, Mitchell SE, Doebley J, Kresovich S, Goodman MM, Buckler ES:玉米关联群体数量性状位点分析的高分辨率平台。植物学报,2005,44(6):1054-1064。
- 6。
Mackay I,Powell W:作物中联动不平衡映射的方法。植物科学趋势。2007,12(2):57-63。
- 7。
Gupta PK,Rustgi S,Kulwal PL:延长植物中的联系不平衡和协会研究:现状和未来前景。植物摩尔巴二醇。2005,57(4):461-485。
- 8。
玉米1号染色体DNA序列多态性研究(英文)Zea Mays SSP。梅斯l .)。美国国家科学院学报。中国科学(d辑),2001,98(16):9161-9166。
- 9。
在一种近交系植物中检测到的极端群体依赖连锁不平衡,Hordeum Vulgare..中国生物医学工程学报。2006,27(1):557-567。
- 10.
Hirschhorn JN,Daly MJ:常见疾病和复杂性状的基因组协会研究。NAT Rev Genet。2005,6(2):95-108。
- 11.
Atwell S, Huang YS, Vilhjalmsson BJ, Willems G, Horton M, Li Y,孟D, Platt A, Tarone AM, Hu TT, et al .: 107个表型的全基因组关联研究拟南芥蒂利亚纳自交系。自然。2010,465(7298):627-631。
- 12.
Thornsberry JM,Goodman MM,Doebley J,Kresovich S,Nielsen D,Buckler ES:Dwarf8多态性与开花时间的变化相关联。自然遗传学。2001,28(3):286-289。
- 13.
acta botanica sinica(云南植物研究中心);acta botanica sinica(云南植物研究中心);植物学报。2003,15(8):1795-1806。
- 14.
Stracke S, Haseneyer G, Veyrieras JB, Geiger HH, Sauer S, Graner A, Piepho HP:关联图谱揭示了决定大麦开花时间的基因作用和互作。acta photonica sinica, 2009, 40(2): 369 - 372。
- 15.
关键词:硬粒小麦,Psy1-A1,等位基因变异,黄色素遗传工程学报。2009,32(8):1539-1548。
- 16。
李艳红,张超,高志松,Smulders MJM,马志林,刘志新,南红艳,常仁志,邱丽娟:大豆孢囊线虫抗性候选基因rhg1的SNP标记及单倍型分析。分子育种。2009,24(1):63-76。
- 17。
陈志强,陈志强,陈志强,等:拟南芥开花时间候选基因关联定位。中国生物医学工程学报。2009,32(1):1 - 8。
- 18。
赵敏,aranzana mj,金斯,侦探c,shindo c,唐克尔,托玛jian c,zheng hg,dean c,marjoram p等,结构化样本中的关联映射的拟南芥例子。Plos遗传学。2007,3(1):
- 19。
Kilian B, Özkan H, Kohl J, von Haeseler A, Barale F, Deusch O, Brandolini A, Yucel C, Martin W, Salamini F: 7个大麦基因的单倍型结构:与基因库瓶颈、穗型系统发育和大麦驯化位点的相关性。分子生物学与分子生物学杂志。2006,27(3):230-241。
- 20。
Morrell PL, Clegg MT:肥沃新月以东大麦(Hordeum vulgare)第二次驯化的遗传证据。中国科学院院刊。2007,104(9):3289-3294。
- 21。
kilian b,Özkanh,pozzi c,萨拉米尼f:驯化Triticeae在新月沃土小麦科的遗传学和基因组学。编辑:Muehlbauer G, Feuillet C. 2009,施普林格纽约,7:81-119。
- 22.
Hayes P,Szucs P:大麦的不平衡和关联:在玻璃外思考。美国国家科学院学报。2006,103(49):18385-18386。
- 23.
Schulte D, Close TJ, Graner A, Langridge P, Matsumoto T, Muehlbauer G, Sato K, Schulman AH, Waugh R, Wise RP, et al: The International Barley Sequencing Consortium—At The Threshold of Efficient Access to The Barley Genome。植物生理学报。2009,149(1):142-147。
- 24.
Rostoks N,Ramsay L,Mackenzie K,Cardle L,Bhat Pr,罗斯ML,Svensson JT,Stein N,Varshney RK,Marshall DF等人:最近的人工交叉历史促进了在精英近亲作物品种中的全基因组关联映射。美国国家科学院学报。2006,103(49):18656-18661。
- 25.
Wenzl P,Li H,Carling J,周M,Raman H,Paul E,Hearnnp,Maier C,XIA L,Caig V等人:将Barley与SSR RFLP和STS Loci连接的高密度共识图。和农业特征。BMC基因组学。2006年,7:206-
- 26.
关闭TJBP Lonardi年代,吴Y, Rostoks N, L,拉姆塞Druka,斯坦N, Svensson JT,沃纳梅克年代,Bozdag年代,吹捧ML, Moscou MJ,曹国伟年代,Varshney RK,深圳űcs P,佐藤K,海耶斯点,马修斯德,Kleinhofs, Muehlbauer GJ, DeYoung J,马歇尔DF Madishetty K,芬顿RD, Condamine P,格兰,沃R:大麦高通量SNP基因分型的开发与实施。中国生物医学工程学报。2009,10:582-
- 27.
Hadeyer G,Stracke S,Piepho HP,Sauer S,Geiger Hh,Graner A:大麦胚乳发展中涉及的转录因子的DNA多态性和单倍型模式与关键的农艺性状有关。BMC植物生物学。2010,10:
- 28。
Cockram J,White J,Leigh FJ,Lea Vj,Chiapparino E,Laurie da,Mackay Ij,Powell W,O'Sullivan DM:Barley中分区基因座的关联映射。BMC Genet。2008,9:16-
- 29。
Ramsay L,Comadran J,Druka A,Marshall DF,Thomas WTB,Macaulay M,Mackenzie K,Simpson C,Fuler J,Bonar N等:中等-C,是大麦小穗侧向育性的修饰因子,是玉米驯化基因的同源基因Teosinte分支1.NAT Genet。2011,43(2):169-172。
- 30.
Waugh R, Jannink JL, Muehlbauer GJ, Ramsay L:大麦全基因组关联扫描的出现。植物学报,2009,12(2):218-222。
- 31。
Michalek Thiel,Graner varshney:利用Barley的基因衍生的SSR标记的开发和表征的利用数据库(Hordeum Vulgare.l .)。标记理论和应用的遗传学。2003,106(3):411-422。
- 32。
王志强,王志强。小麦籽粒大小与籽粒品质的关联映射(Triticum Aestivum L.)品种。遗传学。2006,172(2):1165-1177。
- 33。
Myles S,Peiffer J,Brown PJ,Ersoz Es,Zhang Z,Costich de,Buckler ES:关联映射:关键考虑因素从基因分型转向实验设计。植物细胞。2009,21(8):2194-2202。
- 34.
Vosmer Linden CG,Vosmer B,VAN EEUWIJK FA:使用血统信息与抗马铃薯植物植物的抗性的插图结合映射的混合模型方法。遗传学。2007,175(2):879-889。
- 35.
Pritchard JK, Stephens M, Rosenberg NA, Donnelly P:结构化种群中的关联映射。中国生物医学工程学报。2000,27(1):170-181。
- 36.
Yu J, Pressoir G, Briggs WH, Vroh Bi I, Yamasaki M, Doebley JF, McMullen MD, Gaut BS, Nielsen DM, Holland JB,等:一种统一的混合模型关联映射方法。acta photonica sinica, 2006, 38(2): 203-208。
- 37.
STICH B,MELCHINGER AE:油菜籽,马铃薯,甜菜,玉米和拟南芥相关型映射的混合模型方法比较。BMC基因组学。2009,10:
- 38.
Hadeneyer G,Stracke S,Paul C,Einfeldt C,Broda A,Piepho HP,Graner A,Geiger HH:春灵大麦世界收集的人口结构和表型变异,为协会研究设立。植物育种。2010,129(3):271-279。
- 39.
Knüpffer H, van Hintum T:第13章总结多样性——大麦核心收藏。植物遗传育种进展。编辑:von Bothmer R, van Hintum T, Knüpffer H, Kazuhiro S. 2003, Elsevier, 7: 259-267。
- 40。
Naumann C, Bassler R: VDLUFA-Methodenbuch III: Die Chemische Untersuchung von Futtermitteln, 5。2004, Ergänzungslieferung, Darmstadt, VDLUFA
- 41。
Payne Rw,Murray da,Harding Sa,Baird DB,Soutar DM:Genstat for Windows(第9版)介绍。VSN International,Hemel Hempstead。2006年
- 42。
Fan JB, Gunderson KL, Bibikova M, Yeakley JM, Chen J, Wickham Garcia E, Lebruska LL, Laurent M, Shen R, Barker D: Illumina Universal Bead Arrays。在酶学方法。主编:Alan K, Brian O. 2006,学术出版社,410:57-73。
- 43。
FAN JB,Oliphant A,Shen R,Kermani BG,Garcia F,Gunderson KL,Hansen M,Stemers F,Butler SL,Deloukas P等人:高度平行的SNP基因分型。冷泉港Symp量子Biol。2003,68:69-78。
- 44。
Liu K, Muse SV: PowerMarker:遗传标记分析的集成分析环境。生物信息学。2005,21(9):2128-2129。
- 45。
种群间的遗传距离。国立台湾大学学报(自然科学版),1992,21 (1):1 -
- 46.
基于多位点基因型数据的群体结构推断:连锁位点和相关等位基因频率。中国生物医学工程学报。2003,27(4):427 - 434。
- 47.
Pritchard J, Stephens M, Donnelly P:利用多位点基因型数据推断群体结构。遗传学。2000,155:945-959。
- 48.
Perrier X, Jacquemound-Collet JP: DARwin Software. 2006, [http://darwin.cirad.fr/darwin.]
- 49.
Barrett Jc,Fry B,Maller J,Daly MJ:Haploview:LD和单倍型图的分析和可视化。生物信息学。2005,21(2):263-265。
- 50.
WEIR BS:遗传数据分析II:离散群体遗传数据的方法。1996年,Sinauer Associates,Sunderland,Massachusetts
- 51。
Bradbury PJ,Zhang Z,Kroon de,Casstevens TM,Ramdoss Y,Buckler ES:Tassel:各种样品中复杂性状的关联映射软件。生物信息学。2007,23(19):2633-2635。
- 52。
康慧敏,蔡立伦,王志强,王志强,王志强:模式生物关联图谱中种群结构的有效控制。中国生物医学工程学报。2008,27(3):427 - 434。
- 53。
Stich B,Mohring J,Piepho HP,Heckenberger M,Buckler Es,Melchinger Ae:相关模型方法的比较映射。遗传学。2008,178(3):1745-1754。
- 54。
Benjamini Y,Hochberg Y:控制虚假发现率 - 一种实用而强大的多种测试方法。J Roy Stat SoC B满足。1995,57(1):289-300。
- 55。
Chan Ek,Rowe HC,Kliebenstein DJ:了解防御代谢物的演变拟南芥蒂利亚纳使用基因组关联映射。遗传学。2010,185(3):991-1007。
- 56。
史密斯A,Cullis B,Gilmour A:澳大利亚作物品种评价数据分析。澳大利亚和新西兰统计日报。2001,43(2):129-145。
- 57。
Malysheva-Otto LV,Ganal MW,罗德斯MS:分析分子多样性,人口结构和联系不平衡在全球培养大麦种质调查中的综合(Hordeum Vulgare.l .)。BMC Genet。2006,7:6-
- 58。
张女士,Marchand S,Tinker Na,Belzile F:Barley的群体结构和联动不平衡,Barley通过Dart标记评估。Al Appl Genet。2009,119(1):43-52。
- 59.
Maccaferri M,Sanguineti MC,Noli E,Tuberosa R:杜兰姆小麦精英收集中的人口结构和远程联动不平衡。mol繁殖。2005,15(3):271-290。
- 60。
田晓东:大麦遗传和驯化在全球范围内的重要性。林志刚,王志刚。基于遗传算法的微卫星导航系统设计[j] .光子学报,2007,40(5):691 - 698。
- 61。
Wang G,Schmalenbach I,Von Korff M,Leon J,Kilian B,Rode J,Pillen K:大麦光周期和春化基因与BC开花时间和农艺性状的QTLS协会2DH群体和一套野生大麦渐渗系。应用电子学报。2010,120(8):1559-1574。
- 62。
Laurie Da,Pratchett N,Snape JW,Bezant JH:RFLP映射五个主要基因和冬季X春季大麦(Hordeum Vulgare L.)交叉的开花时间控制开花时间。基因组。1995,38(3):575-585。
- 63。
Faure S, Higgins J, Turner A, Laurie DA: The开花轨迹T在大麦的基因家庭(Hordeum Vulgare.).中国生物医学工程学报,2016,35(1):1 - 8。
- 64。
Comadran J,Russell Jr,展位A,Pswarayi A,Cecarelli S,Grando S,Stanca Am,Pecchioni N,Akar T,Al-Yassin A等,Multi-Envitiopary试验数据的混合模型关联扫描显示重大基因座控制产量地中海环境下的部落恒星产量相关性状。理论和应用的遗传学。2011,122(7):1363-1373。
- 65。
Griffiths S,Dunford RP,Z,Laurie Da:演变君士坦斯像大麦、水稻和拟南芥的基因家族。植物营养与肥料学报。2003,27(4):453 - 457。
- 66。
QI X,Niks Re,STAM P,Lindhout P:鉴定QTL用于大麦叶片生锈(PUCCINIA HORDEI)的QTL。理论和应用的遗传学。1998,96(8):1205-1215。
- 67。
Marqueh-Cedillo La,Hayes Pm,Kleinhofs A,Legge WG,Rossnagel BG,Sato K,Ullrich Se,Wesenberg DM,Proj Nabgm:基于两个精英北美品种的双倍单倍体后代的大麦农艺性状的QTL分析种质群体。理论和应用的遗传学。2001,103(4):625-637。
- 68。
gotwald S, Stein N, Borner A, Sasaki T, Graner A:赤霉素不敏感矮化基因sdw3大麦的染色体位于2HS染色体上,与水稻7L染色体高度共线性。中国生物医学工程学报。2004,27(4):426-436。
- 69。
海耶斯点,刘BH,克纳普SJ,陈F,琼斯B,布雷克T, Franckowiak J, Rasmusson D,索雷尔M,乌尔里希,等:数量性状位点的影响和环境交互北美大麦种质的一个示例。acta photonica sinica, 2011, 40(3): 492 - 498。
- 70.
贾QJ,张XQ,Westcott S,Broughton S,Cakir M,阳JM,Lance R,Li Cd:嗜酸甘油蛋白20-氧化酶基因的表达水平与大麦多种农艺和品质性状有关。理论和应用的遗传学。2011,122(8):1451-1460。
- 71.
YIN X,STAM P,DOURLEIJN CJ,KROPFF MJ:用于春麦片的屈服测定生理特征的定量特质基因座的AFLP映射。理论和应用的遗传学。1999,99(1-2):244-253。
- 72.
关键词:大麦,回交,QTL分析(Hordeum Vulgare.l .)。acta photonica sinica, 2011, 40(2): 362 - 362。
- 73.
关键词:青稞,AB-QTL, qtl分析从野生大麦(H-vulgare ssp spontaneum)渗入农艺性状的有利外来等位基因的检测。遗传工程学报。2006,12(7):1221-1231。
- 74.
Von Korff M,Grando S,Del Greco A,Del Greco A,Baum M,Ceccarelli S:与大麦中的地中海旱地条件相关的定量特质基因座。理论和应用的遗传学。2008,117(5):653-669。
- 75。
Szücs P, Blake VC, Bhat PR, Chao S, Close TJ, custa - marcos A, Muehlbauer GJ, Ramsay L, Waugh R, Hayes PM:大麦连锁图谱和麦芽品质QTL定位的整合资源。植物学报,2009,2(2):134-140。
- 76。
ABDEL-HALEEM H,Bowman J,Giroux M,Kanazin V,Talbert H,Surber L,Blake T:酸性洗涤剂纤维的定量特质和谷物化学成分在覆盖×船体大麦群中。Euphytica。2010,172(3):405-418。
- 77。
Mather De,Tinker Na,Laberge de,Edney M,Jones Bl,Rossnagel BG,Legge WG,Briggs Kg,Irvine RB,Falk de等,影响谷物和麦芽质量的基因组在北美双排中大麦十字架。农作物科学。1997,37(2):544-554。
- 78。
陈凤强,陈凤强,陈凤强:数量性状位点定位在啤酒大麦冬习性发育中的应用。植物育种。1996,115(1):43-51。
- 79。
Rafalski JA:作物改良中的关联遗传。植物生物学进展,2010,13(2):174-180。
- 80.
acta optica sinica, 2010, 31 (4): 494 - 494 . acta optica sinica, 2010, 31 (4): 494 - 494 . acta optica sinica, 2010, 31 (4): 494 - 494 . acta optica sinica, 2010, 31(4): 494 - 494。作物科学,2010,50(2):556-566。
- 81.
Melchinger Ae,Graner A,Singh M,Messmer MM:欧洲大麦种质中的关系.1。RFLP透露冬季和春季品种之间的遗传多样性。农作物科学。1994,34(5):1191-1199。
- 82。
Remington DL,Thornsberry JM,Matsuoka Y,Wilson LM,Whitt SR,Doebley J,Kresovich S,Goodman MM,贝勒斯特:玉米基因组中的连锁不平衡和表型关联的结构。美国国家科学院学报。2001,98(20):11479-11484。
- 83。
Stracke S,Presterl T,Stein N,Perovic D,Ordon F,Graner A:渗入和重组对单倍型结构的影响和围绕麦芽病毒性抗性的轨迹围绕的锁定性膨胀性。遗传学。2007,175(2):805-817。
- 84。
Kraakman At,Niks Re,Van den Berg Pm,STAM P,Van Eeuwijk Fa:在现代春季大麦品种中的产量和产量稳定性的联动不平衡映射。遗传学。2004,168(1):435-446。
- 85。
Comadran J,Thomas Wt,Van Eeuwijk Fa,Ceccarelli S,Grando S,Stanca Am,Pecchioni N,Akar T,Al-Yassin A,Benbelkacem A等:遗传多样性和连锁不平衡在高度结构中的模式Hordeum Vulgare.为地中海盆地绘制人口图。应用电子学报。2009,119(1):175-187。
- 86。
张Z,ersoz e,赖cq,todhunter rj,tiwari hk,gore ma,bradbury pj,yu j,arnett dk,Ordovas jm,等:混合线性模型方法适用于基因组 - 宽协会研究。自然遗传学。2010年,42(4):355-360。
- 87。
Price AL, Patterson NJ, Plenge RM, Weinblatt ME, Shadick NA, Reich D:主成分分析修正了全基因组关联研究中的分层。acta photonica sinica, 2006, 38(8): 904-909。
- 88。
Komatsuda T,Pinkheirandish M,He C,Azhaguvel P,Kanamori H,Perovic D,Stein N,Graner A,柳条T,Tagiri A,等,起源于同型域 - 亮氨酸拉链I-Class的突变Homeobox基因。美国国家科学院学报。2007,104(4):1424-1429。
- 89。
Buckler ES,荷兰JB,Bradbury PJ,Acharya CB,Brown PJ,Browne C,Ersoz E,Flint-Garcia S,Garcia A,Glaubitz JC等:玉米开花时间的遗传建筑。科学。2009,325(5941):714-718。
- 90。
张j,李z,张ch:矮化基因分析Zhepi 1.和Aizao 3:中国大麦育种中的两位矮化基因供体。植物学报,2007,87(1):93-96。
- 91.
Kleinhofs A:集成大麦RFLP和古典标记图。大麦类型新闻信。1997年,27:105-112。
- 92.
王志强,王志强,王志强:小麦蜡质位点的结构分析。中国生物医学工程学报,1998,16(4):591 - 598。
- 93.
Manolio TA, Collins FS, Cox NJ, Goldstein DB, Hindorff LA, Hunter DJ, McCarthy MI, Ramos EM, Cardon LR, Chakravarti A, et al:复杂疾病缺失遗传性的发现。自然。2009,461(7265):747-753。
- 94.
Roy JK,Smith Kp,Muehlbauer GJ,Chao S,Close TJ,Steffenson BJ:野生大麦斑块斑块抗性的关联映射。mol品种。2010,26(2):243-256。
- 95.
Maher B:个人基因组:遗传性缺失的案例。自然。2008,456(7218):18-21。
- 96.
Visscher PM:估算人类身高变化。中国生物医学工程学报。2008,40(5):489-490。
- 97.
Frazer Ka,Murray Ss,Schork NJ,Topol EJ:人类遗传变异及其对复杂性状的贡献。NAT Rev Genet。2009,10(4):241-251。
- 98.
Gibson G:GWAS中隐藏遗传性的提示。NAT Genet。2010年42日(7):558-560。
- 99。
HALL D,TEGSTROM C,INGVARSOSON PK:使用关联映射来描述植物中复杂性状的遗传基础。简短的Funct基因组学。2010,9(2):157-165。
- One hundred.
Blott S,Kim Jj,Moisio S,Schmidt-Kuntzel A,Cornet A,Berzi P,Cambisano N,Ford C,Grisart B,Johnson D等,定量性状轨迹的分子解剖:苯丙氨酸 - 酪氨酸替代物在牛脑结构域的牛生长激素受体与对奶产率和组成的重大影响有关。遗传学。2003,163(1):253-266。
- 101。
Brachi B,Faure N,Horton M,Flahauw E,Vazquez A,Nordborg M,Bergelson J,Cuguen J,Roux F:rapidopsis Thaliana开花时间的联系和关联映射性质。Plos Genet。2010年,6:E1000940-
- 102。
Mott R,Flint J:使用异构股票的数量特质基因座的同时检测和精细映射。遗传学。2002,160(4):1609-1618。
- 103。
Mayer KFX, Martis M, Hedley PE, Simkova H, Liu H, Morris JA, Steuernagel B, Taudien S, Roessner S, Gundlach H,等:利用染色体和比较基因组学破解大麦基因组。植物学报。2011,23(4):1249-1263。
- 104。
Cockram J,White J,Zuluaga DL,史密斯D,Comadran J,Macaulay M,Luo Zw,Kearsey MJ,Werner P,Harrap D等,以候选人的大麦基因组中的候选多态性分辨率。美国国家科学院学报。2010,107(50):21611-21616。
确认
我们感谢Kathrin Baake、Kerstin Wolf、Ute Krajewski、Jürgen Marlow和Peter Schreiber提供的出色技术援助。特别感谢Nils Stein和Kerstin Neumann的有益讨论。我们要感谢GABI-GENOBAR联盟的所有合作伙伴,特别是Stephan Weise提供的生物信息学支持。我们还感谢GABI-GENOPLANT合作伙伴在实地试验中提供的支持。这项工作是由德国教育和研究部(BMBF)的GABI项目资助的。
作者信息
隶属关系
通讯作者
附加信息
作者的贡献
RKP进行了研究并执行了数据固化,关联分析,对数据的解释并起草了稿件。RS参与统计分析和改进手稿。MM和FAE建议统计分析。GH生成了表型数据。BK协调该项目,并协助戈尔登特数据分析并改进稿件。AG启动了该研究,参与了数据解释和改进稿件。所有作者都读过了最后的手稿。
电子补充材料
12870 _2011_1003_moesm1_esm.xlsx
附加文件1:表S1来自IPK定制OPA的957个SNP标记的信息,这些标记在我们的小组中是成功的。(XLSX 68 KB)
12870_2011_1003_moesm2_esm.xlsx.
附加文件2:表S2用于GWAS的212种附加的详细信息。登录,行类型,成功标记数,结构组,原产地区和原产国的名称。(XLSX 19 KB)
图S1
附加文件3:使用DArT标记结构结果。日志概率数据(LNP(D))作为功能K.(群集数量)来自结构运行的使用1088 DART标记,具有相同的关联面板。图表的高原K= 6表示面板中可能的最小子组数。(JPEG 18 KB)
12870_2011_1003_moesm4_esm.jpeg.
附加文件4:图S2表型分布为224个弹簧大麦的分布,用于特征标题日期(HD),植物高度(PHT),千粒重(TGW),淀粉含量(SC)和蛋白质含量(CPC)。(JPEG 469 KB)
12870 _2011_1003_moesm5_esm.docx
额外文件5:表S3两排和六排组的表型变异。估计两轮和六轮组中每个性状的均值、标准差(SD)、变异(VAR)、标准误差变异(SEVAR)和变异系数(CV%)。(多克斯21 KB)
12870_2011_1003_moesm6_esm.docx.
附加文件6:表S4在面板中所有六个子组中的平均值,SD,变化(var),标准误差变化(sevar),标准误差变化(sevar)和方差系数(cv%)。(DOCX 29 KB)
12870_2011_1003_moesm7_esm.jpeg.
附加文件7:图S3 BLUPs和BLUEs的淀粉含量比较从图中可以看出,在我们的实验中,BLUPs和BLUEs并没有太大的区别。(JPEG 410 KB)
12870_2011_1003_moesm8_esm.xlsx.
附加文件8:表S5在面板中GWA中使用的918 SNP标记的标记多态性信息。(XLSX 60 KB)
12870_2011_1003_moesm9_esm.jpeg.
附加文件9:图S4基于使用918 snp衍生的前两个成分的面板主坐标分析(PCoA)。初生轴倾向于根据穗的形态特征分成亚群(蓝色:六棱大麦;红色:而且大麦)。进一步的聚类是基于资源的来源。(JPEG 940 KB)
图S5
附加文件10:大麦中每条染色体的LD图。正方形的颜色说明了成对的强度r2黑白刻度上的值,黑色表示完美的LD(r2= 1.00),白色表示完全平衡(r2= 0)。不考虑失败、单型SNPs以及MAF < 0.05的SNPs。(JPEG 9 MB)
12870 _2011_1003_moesm11_esm.jpeg
附加文件11:使用不同关联模型(天真,P,Q,QK,PK和K)的行类型的图S6 GWAS全基因组扫描。(JPEG 584 KB)
12870 _2011_1003_moesm12_esm.jpeg
附加文件12:所有特征的图S7 GWA。QTL的定位和候选基因的特征行型(RT),标题(HD),植物高度(PHT),千粒重(TGW),淀粉含量(SC)和粗蛋白质含量(CPC)的遗传图谱有918个SNP标记。(JPEG 5 MB)
作者的原始提交的图像文件
权利和权限
开放获取本文在“生物资源”中央有限公司的许可下公布了这是一个开放式访问条款,分配根据创意公约归因许可证的条款(https://creativecommons.org/licenses/by/2.0.),允许在任何媒介上无限制地使用、分发和复制,但必须正确引用原作。
关于这篇文章
引用这篇文章
PASAM,R.K.,Sharma,R.,MaloSetti,M。等等。全球春季大麦集邮农艺性状的基因组协会研究。BMC植物杂志12日,16(2012)。https://doi.org/10.1186/1471-2229-12-16
收到:
接受:
发表:
关键词
- 定量特质基因座
- 连锁不平衡
- 多态信息含量
- 数量性状位点区
- 粗蛋白质含量